De nouveaux réseaux booléens logiques pour décrire le comportement des cellules
Si l’on comprend les grands principes de certains phénomènes biologiques, il faut pouvoir les modéliser même lorsque leurs détails restent mal connus. Les réseaux booléens remplissent cette fonction depuis des décennies, mais ils se trompent parfois et se limitent à des systèmes réduits. Des chercheurs ont proposé un nouveau type de réseau booléen, basé sur une permissivité maximale afin de ne manquer aucune possibilité. Paradoxalement, leur système est moins gourmand en puissance de calcul et pourrait s’appliquer à des cas aussi vastes qu’un génome entier.
La prédiction de processus biologiques complexes, comme la division cellulaire, demande de prendre en compte un grand nombre d’éléments dont nous possédons une connaissance globale, mais dont le détail nous échappe encore trop souvent. Les réseaux booléens, issus de la logique du même nom où les composants n’ont que deux états possibles (vrai ou faux), ont été introduits en biologie dans les années 60 pour faire face à ces difficultés.
« Les réseaux booléens permettent de modéliser les comportements des cellules, en fonction de l’expression et de l’activité de leurs gènes, explique Loïc Paulevé, chargé de recherche CNRS au Laboratoire Bordelais de Recherche en Informatique (LaBRI - CNRS/Université de Bordeaux/Bordeaux INP). On suit alors de façon logique l’activation des gènes et des protéines selon différentes conditions, grâce au langage formel, sans avoir à décrire la nature exacte de leurs interactions. On passe d’un système complexe à un objet mathématique simple, qui colle mieux au niveau de connaissance que nous avons réellement des systèmes biologiques. »
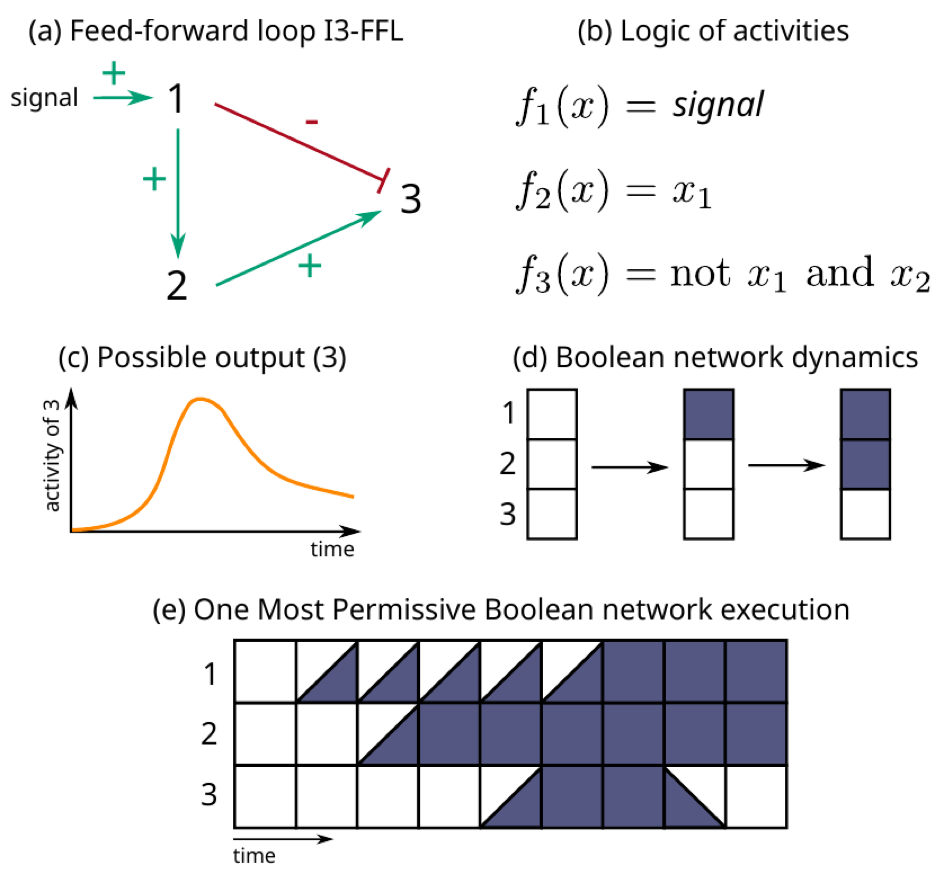
Si ces objets mathématiques sont efficaces et aisés à définir, l’analyse de leur dynamique est cependant souvent très gourmande en temps de calcul, et reste limitée à des réseaux d’une centaine de composants. Dans un article publié dans la revue Nature Communications, Loïc Paulevé et ses collègues ont montré que les réseaux booléens, tels qu’ils étaient jusqu’alors utilisés, pouvaient aboutir à de conclusions imprécises pouvant mener à des erreurs d'interprétation sur la dynamique du système. Ils manquent en effet certaines possibilités lorsque certaines interactions agissent sur plusieurs échelles de temps (voir fig.). Ajouter à ces réseaux tous les détails nécessaires pour qu’ils puissent y faire face les surchargerait rapidement. Ces chercheurs du LaBRI et du Laboratoire Spécification et Vérification (LSV - CNRS/ENS Paris-Saclay) ont surtout rendu ces systèmes plus performants grâce à une nouvelle méthode.
Les chercheurs ont pour cela introduit le concept de Most Permissive Boolean Networks (MPBN). Celui-ci prédit, à partir de la logique du réseau booléen et sans paramètre supplémentaire, toutes les évolutions permises de leurs états, quelles que soient leurs caractéristiques temporelles et quantitatives. Si un MPBN ne parvient pas à reproduire une observation faite sur des cellules, aucun modèle compatible, même intégrant du temps ou des quantités, ne le peut. Les chercheurs l’ont par exemple testé sur des modèles de prolifération de tumeurs et de différenciation de lymphocytes T.
Il existe deux méthodes classiques pour calculer les comportements possibles d’un réseau booléen : la mise à jour synchrone et la mise à jour asynchrone. Dans le premier cas, tous les composants du modèle changent d’état à la fois, contre un seul dans le second. Les réseaux booléens asynchrones sont les plus employés, mais ils ratent parfois des comportements car ils ne tiennent pas compte des différents seuils et temps d’interactions.
Les MPBN parviennent à prédire davantage de comportements que ces deux méthodes, tout en évitant l’explosion du nombre d’états et de temporalités à gérer en même temps dans le modèle. En plus de ne pas manquer d’évènements, les MPBN demandent beaucoup moins de puissance de calcul et peuvent ainsi décrire des systèmes plus vastes et complexes. Alors qu’un réseau booléen asynchrone se limite généralement à moins d’une centaine de composants, les chercheurs ont fait fonctionner un réseau simulé atteignant les 100 000 composants.
En plus de cette publication, Loïc Paulevé a développé un logiciel permettant d’utiliser des MPBN. L’analyse des comportements engendrés par les réseaux booléens les plus permissifs ouvre, en particulier, la voie à la vérification et à la synthèse de réseaux à l’échelle du génome entier, avec plusieurs dizaines de milliers de composants.
Référence :
Loïc Paulevé, Juraj Kolčák, Thomas Chatain & Stefan Haar. Reconciling qualitative, abstract, and scalable modeling of biological networks. Nature Communications, volume 11, Article number: 4256 (2020).